单细胞RNA测序(scRNA-seq)技术的发展为我们揭示眼科学中的细胞类型、细胞发育和分化过程以及细胞间相互作用等重要生命现象提供了新的研究视角。然而,在眼科领域进行单细胞全长转录组研究时仍面临一些挑战。传统的单细胞转录组技术主要采用基于短读长的RNA测序方法,虽然可以定量描述基因表达水平,但无法精细区分基因不同异构体。而在眼科领域,了解RNA可变剪切在眼发育、分化以及眼疾病发生发展等过程中的关键调控作用至关重要。
为了解决这些问题,单细胞全长转录组技术应运而生。这些新一代测序技术,如PacBio和Nanopore,可以直接测序全长转录本,从而提供更全面的转录信息。然而,目前该技术在眼科领域的应用还相对较少,主要是由于低通量和高测序错误率等限制,通常只能处理少量细胞,限制了对眼科组织中多种细胞类型的全面分析,也无法捕捉到低丰度的转录本信息,极大限制了该技术的应用。因此,推动单细胞全长转录组技术的发展将为我们更好地理解眼部组织的细胞异质性、发育过程以及疾病机制提供重要的技术手段。

最近,中山大学中山眼科中心刘奕志/肖传乐团队于2023年5月6日在Nature Communications杂志上联合发表题为“High-throughput and high-accuracy single-cell RNA isoform analysis using PacBio circular consensus sequencing”的研究论文,该项研究通过多个RNA链接建库,建立了高通量的全长单细胞RNA测序方法(HIT-scISOseq),在保持测序精度前提下,突破性将单细胞全长RNA测序的通量提高到了一个新水平。
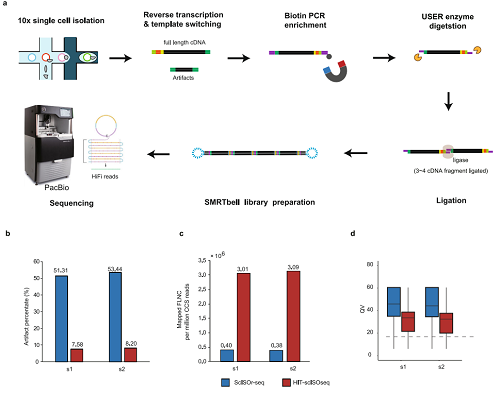
图1.HIT-scISOseq实验流程示意图和技术效果
该研究应用HIT-scISO-seq对食蟹猴角膜缘组织细胞进行分析,在RNA异构体水平,高度重现了已知的细胞类型特征(图2),同时也发现了大量细胞特异的marker isoform,以及揭示了角膜缘组织细胞特异的RNA异构体剪切和表达模式。总的来说,这项研究建立的HIT-scISOseq技术,能够高通量、高精度研究单细胞转录异构体,提供更全面的转录特征信息,极大提高了研究者们在各种生物学过程中开辟新的科学发现的可能性。
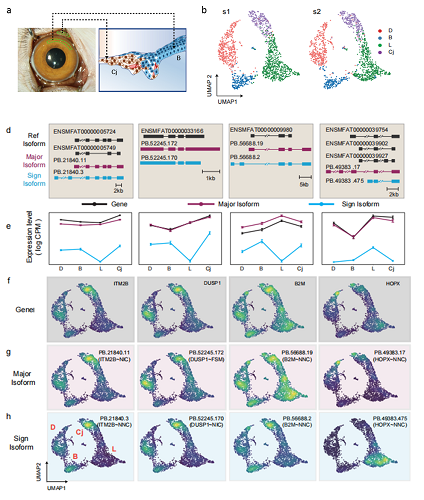
图2.食蟹猴角膜缘组织单细胞RNA异构体表达图谱
HIT-scISOseq技术的引入会为眼科学单细胞研究带来重要进展。该技术的创新性在于通过连接技术结合PacBio HiFi长读长的测序优势,实现了单细胞全长RNA测序的高通量化,显著提高了数据的可靠性和全面性。在食蟹猴角膜缘组织细胞解析中的应用进一步证明了该技术在眼科学基础研究和临床研究中的潜力。HIT-scISOseq技术是一项通用的技术,能够在未来的眼部疾病研究中,对眼部组织中不同细胞类型的RNA异构体进行全面的分析,深入了解疾病的发病机制和分子调控网络以及发现潜在的治疗靶点,为靶向治疗和新药开发提供新的方向和目标。